近日,电子信息与电气工程学院自动化系沈红斌教授、袁野副教授的课题组(模式识别与生物信息学课题组)在《自然-计算科学》(Nature Computational Science)上发表题为《利用图神经网络对空间转录组数据进行细胞聚类》(Cell clustering for spatial transcriptomics data with graph neural networks)的研究论文。

★ 论文简介★
空间转录组技术是生物信息学领域近年来的重大突破之一。该技术通过同时测量大量细胞的空间位置和细胞内的转录组计数,弥补了单细胞测序技术难以测量单个细胞之间位置关系的缺陷,从而为理解多细胞之间的相互作用提供了全新的数据基础。发展针对空间转录组数据的基础分析方法是当前生物信息学领域的前沿问题之一。
论文提出了一种基于图卷积神经网络的空间转录组细胞聚类方法(Cell Clustering for Spatial Transcriptomics,CCST)。此前针对该类数据的处理模型大都基于“同种细胞在空间上相互临近”的这一假设。相比之下,CCST并不依赖于局部特征,而是可以从细胞的全局空间分布中学习节点嵌入。具体方案为:首先将空间转录组建模为图结构的数据。图中每一个节点代表一个细胞,其原始表征为高维的基因表达信息。图的邻接矩阵则依据细胞之间的空间距离建立。随后分别提取图上的局部特征和全局特征,以最大化局部和全局特征间的互信息为目标,训练图神经网络模型,从而得到带有全局结构信息的节点嵌入,再对各细胞所对应的表示向量进行聚类来识别细胞类型,最后从生物功能角度对聚类结果进行了深入分析。
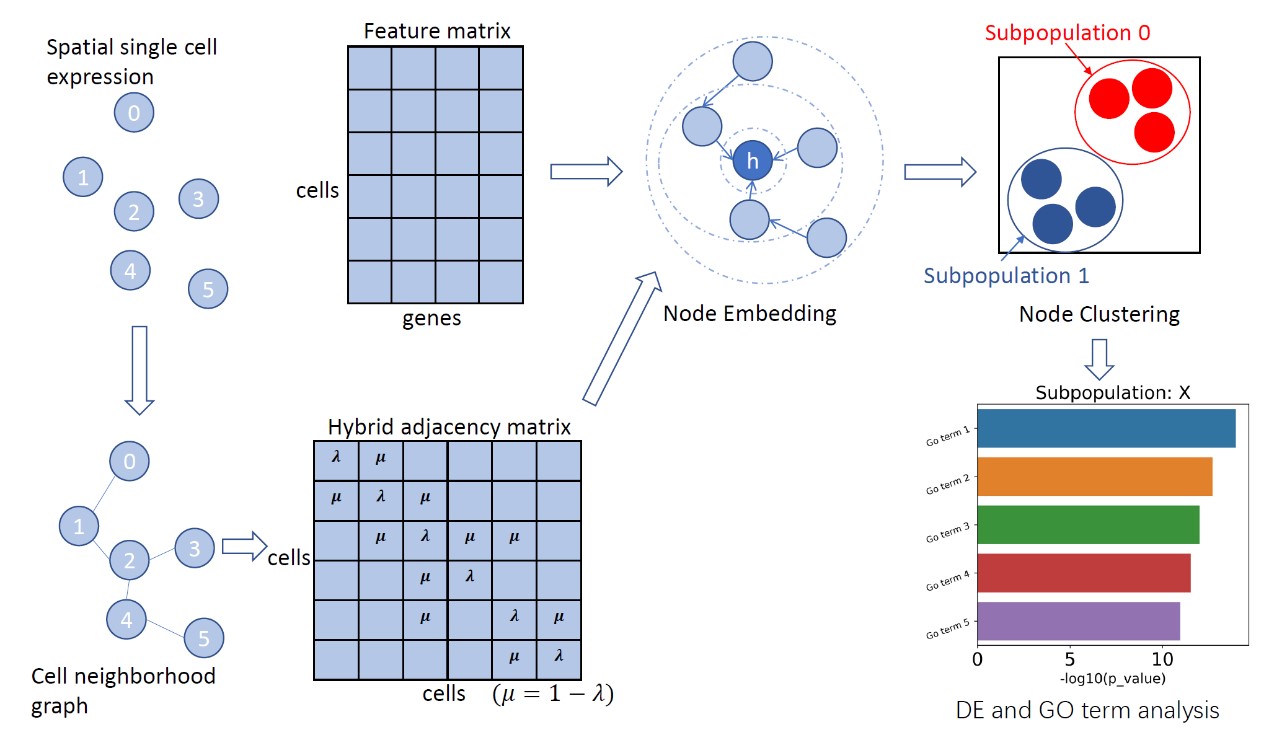
CCST方法示意图
★ 创新之处★
论文在几种不同类型数据集上,综合对比了近期学术界提出的多个相关算法。在脑前额叶皮质和人乳腺癌细胞ST数据集上的实验表明,CCST在ARI、NMI和FMI等多个聚类评价指标上均体现出优势。同时,此方法得到的聚类结果具有较好的生物可解释性。在MERFISH数据中,通过进行差异表达基因和基因本体(GO)分析,可以发现使用CCST聚类出的各个细胞簇能够很好地和各细胞周期阶段对应。
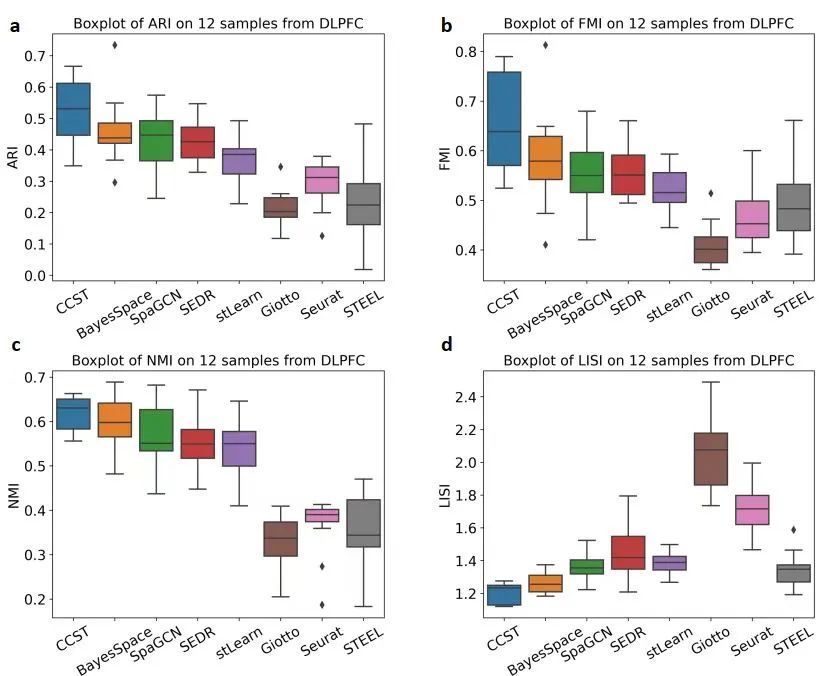
CCST与相关算法的指标对比:a. 调整兰德系数(ARI);b. 归一化互信息(NMI);c. Fowlkes-Mallows分数(FMI);d. 局部逆辛普森指数(LISI)
此研究为处理空间转录组数据提供了新方案,具有被应用于生命医药科学中多层次基础问题研究的潜力,包括建模基因表达的空间分布、分析细胞动力学以及发现关键细胞亚型相互作用及其分子机制等;同时该研究也具有潜在的广泛医学应用场景。
★ 作者信息★
电院自动化系博士生李家琛为论文的第一作者,电院沈红斌教授、袁野副教授为通讯作者,潘小勇助理教授和陈思衡副教授为共同作者。该研究获得国家自然科学基金项目和上海浦江计划的资助。
★ 关于Nature Computational Science★
Nature Computational Science(《自然-计算科学》)是专注于基础和应用计算科学的《Nature》子刊,侧重于计算技术和数学模型的开发与使用,以及它们在解决一系列科学学科中复杂问题的应用。该杂志的主要目标是促进多学科研究和新计算技术的跨学科应用。
论文链接
https://www.nature.com/articles/s43588-022-00266-5?utm_source=natcomputsci_etoc&utm_medium=email&utm_campaign=toc_43588_2_6&utm_content=20220628
本文转载于上海交大电院官微